
Описание компьютерной программы Protein 3D. Е.Л. Демченко, В.А. Карасев (http://Protein-3d.ru)
.docxОбработка результатов
Цель работы:
Знакомство с компьютерной программой «PROTEIN 3D» и ее использование для анализа белковых структур.
Описание компьютерной программы «Protein 3D»
Компьютерная программа «Protein 3D» (Визуализатор надмолекулярных биоструктур «Protein 3D») является одним из таких визуализаторов, предназначенных для просмотра файлов в формате PDB. Помимо различных и общепринятых форм рендеринга, в программе реализованы представления о системах сопряженных ионно-водородных связей, которые делают ее в своем роде уникальной и полезно для изучения студентами.
Учебное задание №1.
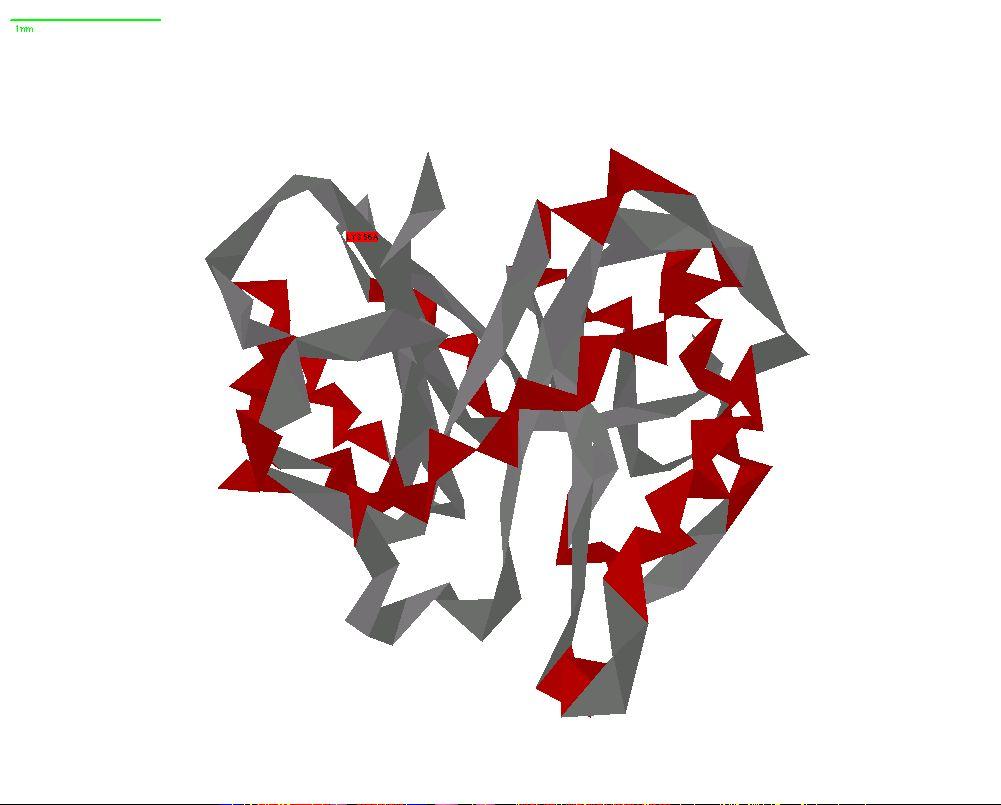
На белом фоне в форме представления Atoms 1 (иконка Render) с качеством Best Quality в формате .jpeg и производят запись картинки в файл и с помощью Windows проверяют качество записи.
1)При помощи иконки «file» открываем файл «4HHB.PDB»
Содержание всех команд, представленных в иконке File, показано на рис. 1.

Рис. 1. Команды, записанные в иконке File.
2) С помощью иконки «Render» меняем форму представления структуры анализируемого объекта на «atom 1».
В иконке Render содержатся команды, обеспечивающие различные формы представления (рендеринга) структуры анализируемых объектов. Мы проведем разбор этих форм на белках.

Рис 2. Содержание команд иконки «Render».
3) Меняем качество рисунка на «Best quality» во вкладке «JPEG File quality» в иконке «File».
4)Сохраняем результат в формате «jpeg» при помощи иконки «File».

Учебное задание 2.
Записать выделенные в субъединице гемоглобина ССИВС в текстовый файл и проверить наличие записи в файл. Студенты все выполняют предложенное задание. В результате должен быть записан текстовый файл с названием 4HHB.txt.
1) Для начала поставим во вкладке «Select bond types» иконки «CIHBS» галочку на против «Show all».
Иконка CIHBS (сокращение от Conjugated Ionic-Hydrogen Bonвs Systems - Системы Сопряженных Ионно-Водородных связей – ССИВС) содержит ряд специально разработанных команд для работы с этими системами.
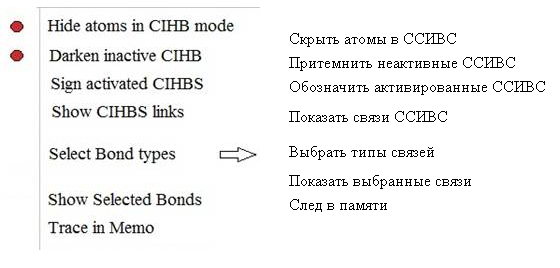
Рис 4. Команды иконки CIHBS.
Команды «Скрыть атомы в ССИВС» (Hide atoms in CIHB mode) и «Притемнить неактивные ССИВС» (Darken inactive CIHB) отмечены красными кружками. В программе они являются установленными заранее и предназначены для улучшения восприятия выделяемых в анализируемой структуре. ССИВС. Так, в отсутствие команды «Hide atoms in CIHB mode», ряд атомов, в первую очередь атомы углерода боковых цепей, создают дополнительный «шум» и затрудняют просмотр ССИВС. Включение этой команды существенно улучшает восприятие ССИВС.
Команды «Обозначить активированные ССИВС» («Sign activated CIHBS») и «Показать связи ССИВС» («Show CIHBS links»). Для анализа ССИВС важно знать, между какими атомами такие связи образовались. Эта информация реализуется с помощью команд «Sign activated CIHBS» («Обозначить активированные ССИВС») и «Show CIHBS links» («Показать связи ССИВС»).
2)Затем поставим галочку напротив «Trace in Memory» в иконке «CIHBS».
Эта установка позволяет в дальнейшем производит запись информации в текстовый файл.
3) Далее нажмем на вкладку «Show CIHBS links» в иконки «CIHBS».
Ниже приведены все вязи ССИВС из файла 4HHB.txt
PGH 249A N 230A O ; System 1 D=3.630
PGH 249A N GLU 165A OE2 ; System 1 D=2.665
PGH 249A N GLU 165A OE1 ; System 1 D=3.423
PGH 249A O2 HIS 95A NE2 ; System 1 D=2.659
PGH 249A O2 ASN 10A ND2 ; System 1 D=3.784
PGH 249A O3 232A N ; System 1 D=3.614
PGH 249A O3 LYS 12A NZ ; System 1 D=3.552
GLU 165A OE1 CYS 126A SG ; System 1 D=3.761
231A N 8A O ; System 1 D=2.831
GLU 97A OE1 LYS 12A NZ ; System 1 D=3.684
PGH 249A O1 GLU 165A OE2 ; System 1 D=3.120
PGH 249A O6 232A N ; System 1 D=2.631
GLU 97A OE2 LYS 12A NZ ; System 1 D=2.597
PGH 249A P PGH 249A O6 ; System 1 D=1.434
231A O 10A N ; System 1 D=2.707
PGH 249A O6 233A N ; System 1 D=3.616
PGH 249A P PGH 249A O4 ; System 1 D=1.530
PGH 249A P PGH 249A O5 ; System 1 D=1.471
PGH 249A O4 169A O ; System 1 D=3.586
PGH 249A O4 211A N ; System 1 D=2.762
PGH 249A O4 171A N ; System 1 D=2.789
SER 211A OG 169A O ; System 1 D=2.623
170A N 166A O ; System 1 D=3.536
SER 235A OG 231A O ; System 1 D=3.372
SER 235A OG 10A O ; System 1 D=3.579
SER 211A OG 174A N ; System 1 D=3.501
SER 211A OG 173A N ; System 1 D=2.556
SER 211A OG 172A N ; System 1 D=3.688
167A N 129A O ; System 1 D=2.623
39A O 9A N ; System 1 D=2.541
CYS 41A SG 9A O ; System 1 D=3.788
41A N 9A O ; System 1 D=2.692
41A O 11A N ; System 1 D=2.988
GLN 64A NE2 41A O ; System 1 D=2.841
GLN 64A NE2 43A N ; System 1 D=3.369
GLN 64A NE2 42A O ; System 1 D=3.513
60A O 40A N ; System 1 D=2.924
62A N 40A O ; System 1 D=2.970
GLN 64A NE2 12A N ; System 1 D=3.063
GLN 64A NE2 12A O ; System 1 D=2.847
90A N 61A O ; System 1 D=3.581
122A N 89A O ; System 1 D=2.769
89A N 61A O ; System 1 D=2.694
Учебное задание 3.
Выделить в субъединице гемоглобина ССИВС Н-связи типа NiH…Oi-4, записать их в текстовый файл и проверить наличие записи в файле.
1)ставим в малой иконке галочку против «NiH…Oi-4», что соответствует основной Н-связи в -спирали белка и снимают галочку на «Show all».
Происходит пересчет структуры и при повороте структуры выявляется расположение выделенного типа Н-связей - NiH…Oi-4
2) строку Show selected bonds. Появляется табличка, в которой нажимаем ОК. Появляется плакетка с выделенными связями.
3)Для записи в текстовый файл нажимаем кнопку Save links и в появляющемся окне вписываем название файла с цифрой 1 – 4HHB-1. Записывается тестовый файл 4HHB-1.txt.
Содержание сохраненного файла выглядит следующим образом:
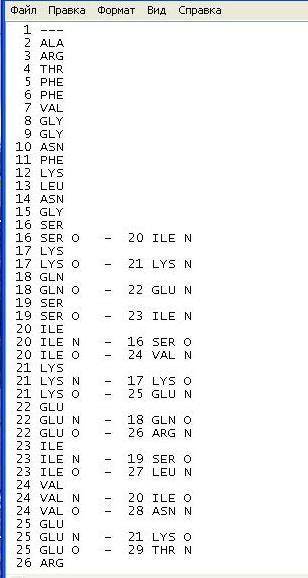
Рис 6. связи типа NiH…Oi-4
Самостоятельная работа
Задание 1:
-
Установить, какой тип структуры является преобладающим в каждом из белков.
-
Установить, имеется ли в предложенных белках кофактор.
-
Установить, связаны ли кофакторы (если они имеются) со структурой ССИВС субъединицы.
-
Установить, состоят ли из белки из субъединиц.
-
а)в белке 7TIM преобладающей является структура серого цвета.
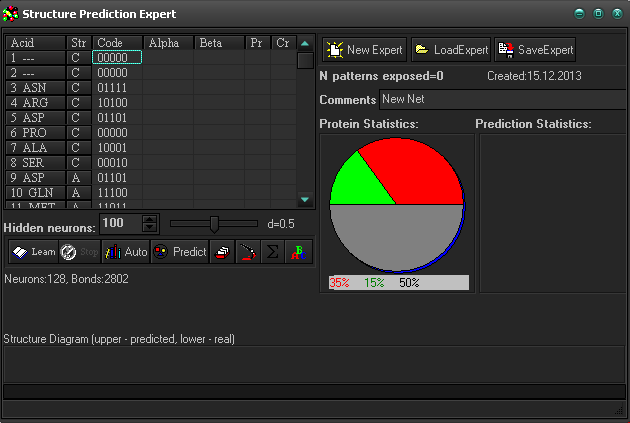
Рис. 7. Прогноз ожидаемой структуры белка 7TIM.
б) в белке ECO RV ENDONUCLEASE (E.C.3.1.21.4) COMPLEXED WITH SUBSTRATE 1 не удалось определить преобладающую структуру. Предположительно из за того что в файле не нанесены данные о структурах (рис. 8)
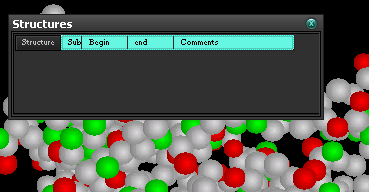
Рис. 8. Данные о структурах белка ECO RV ENDONUCLEASE.
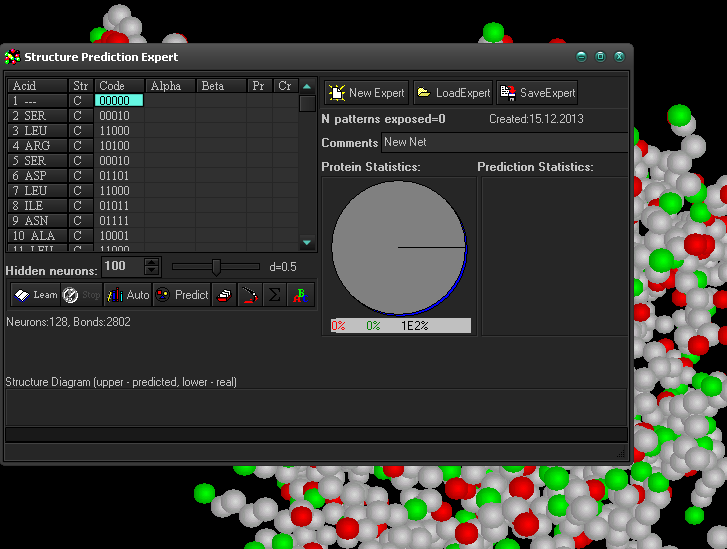
Рис. 9. Прогноз ожидаемой структуры белка ECO RV ENDONUCLEASE.
-
а) в белке CATALASE имеется кофактор.

Рис. 10. кофактор белка 7TIM.
б) в белке ECO RV ENDONUCLEASE кофактор отсутствует.
3. В белке 7TIM присутствуют ССИВС связанные с кофактором.
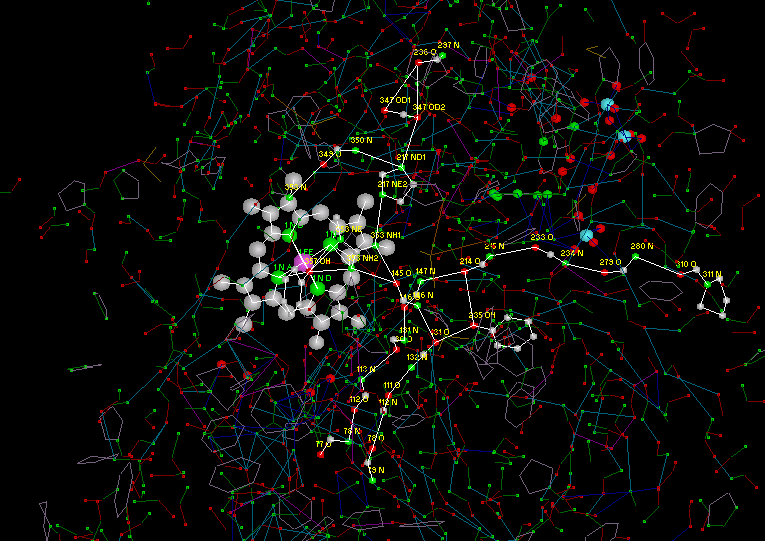
Рис. 11. ССИВС связанные с кофактором в белке 7TIM.
4. а)В белке 7TIM присутствует 2 субъединицы.
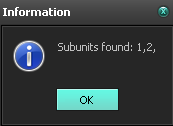
Рис.12. число субъединиц белка 7TIM
б)в белке ECO RV ENDONUCLEASE присутствует 4 субъединицы.
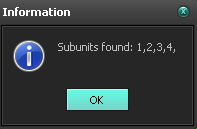
Рис.13. число субъединиц белка ECO RV ENDONUCLEASE
Практически необходимо выполнить следующее:
1. Сделать принт-файлы для каждого из двух белков на белом фоне в рендеринге <PAPER>
2. На белке, содержащем кофактор (ГЕМ – это тоже кофактор) выделить ССИВС, связанные этим кофактором.
3. Получить текстовый файл с описанием выделенной ССИВС
1. принт-файл для белка CATALASE на белом фоне в рендеринге <PAPER>.

принт-файл для белка ECO RV ENDONUCLEASE на белом фоне в рендеринге <PAPER>.
|
№ |
Код белка |
Наименование белка из PDB-файла |
Объем полученного txt-файла |
|
1 |
7TIM |
triosephosphateisomerase |
2 кБ |
|
2 |
1RVA |
ECO RV ENDONUCLEASE (E.C.3.1.21.4) COMPLEXED WITH SUBSTRATE 1 2 DNA |
6 кБ |
