
ИСПОЛЬЗОВАНИЕ ДНК-ТЕХНОЛОГИЙ В МЕДИЦИНЕ - МЕТОДЫ ПЦР И СЕКВЕНИРОВАНИЯ. ГЕННАЯ ТЕРАПИЯ
.pdf
Рис. 20 Принцип метода Сэнгера.
Удлинение цепи ДНК ферментом происходит до момента включения ddNTP. Разделение полученных фрагментов методом электрофореза в геле позволяет определить последовательность нуклеотидов.

Помимо этих компонентов, в реакционную смесь добавляются 4
соответствующих ddNTP (ddA, ddT, ddG и ddC) в концентрациях примерно в 20
раз меньше, чем dNTP. Каждый ddNTP помечен своим флуоресцентным красителем, что позволяет производить анализ в одной пробирке. Ранее для этой цели использовали изотопы P32, а реакцию проводили в 4-х отдельных пробирках для каждого азотистого основания, как и в методе Максама-
Гилберта.
В ходе ферментативного синтеза ДНК, в каком-то из положений случайным образом происходит включение в строящуюся цепь меченного ddNTP вместо обычного dNTP, что приводит к остановке синтеза, так как отсутствие 3ʹ-ОН группы блокирует образование фосфодиэфирной связи со следующим нуклеотидом. Реакцию проводят в циклическом режиме (как при ПЦР). Так как ddNTP составляют примерно 5% от dNTP, а мечение включает
40-50 циклов, в конце такой линейной амплификации получается набор одиночных цепей ДНК, отличающихся по длине и всегда заканчивающихся меченным нуклеотидом (Рис. 20).
После мечения проводят разделение полученных одноцепочечных фрагментов методом электрофореза в ПААГ. В настоящее время данный процесс полностью автоматизирован. Автоматические секвенаторы позволяют проводить электрофорез меченных фрагментов ДНК в тонком капилляре,
заполненном гелем (Рис. 21).
Рис. 21 Капиллярный автоматический секвенатор ABI 3130xl Applied Biosystems (США)
Детекция разделенных фрагментов происходит на дальнем конце капилляра за счет регистрации флуоресценции терминальных ddNTP,
проходящих через детектор фрагментов ДНК. В зависимости от типа терминального ddNTP прибор регистрирует флуоресценцию в той или иной
области спектра. Данные спектрограммы и прочтение нуклеотидной
последовательности отображаются на экране компьютера (Рис. 22).
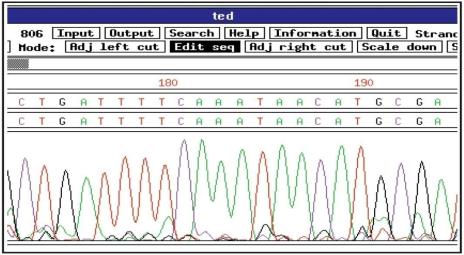
Рис. 22 Спектрограмма, полученная в результате секвенирования по методу Сэнгера на
автоматическом секвенаторе
Анализ данных капиллярного секвенирования сводится к "прочтению"
последовательных пиков флуоресценции. В настоящее время, с использованием современных автоматических секвенаторов, длина одного прочтения по методу Сэнгера составляет 800-1000 нуклеотидов.
2.3 Метод гибридизации на твердой фазе
 В конце 1980-х был предложен подход к определению последовательностей ДНК, получивший название "секвенирование путем гибридизации" (sequencing by hybridization, SBH) или секвенирования на чипе.
В конце 1980-х был предложен подход к определению последовательностей ДНК, получивший название "секвенирование путем гибридизации" (sequencing by hybridization, SBH) или секвенирования на чипе.
Метод основан на гибридизации меченных флуоресцентными метками одноцепочечных фрагментов ДНК с синтетическими олигонуклеотидами известной структуры и длины, которые точечно расположены на твердой основе. При этом на подложке присутствуют все возможные варианты последовательности олигонуклеотида заданной длины. Например, для олигонуклеотида длиной в 8 оснований будут возможны 48=65 536 вариантов последовательностей. Условия гибридизации подбирают таким образом, чтобы только полностью комплементарные фрагменты ДНК взаимодействовали с олигонуклеотидом на подложке.

Рис. 23 Принцип метода секвенирования на чипе
После удаления несвязавшихся молекул ДНК можно зарегистрировать сигнал флуоресценции в тех участках чипа, где находится олигонуклеотид,
комплементарная последовательность которого есть в секвенируемом образце ДНК. Полученный гибридизационный паттерн можно использовать для восстановления исходной последовательности путем сборки перекрывающихся участков сработавших проб (Рис. 23).
К сожалению, невозможно подобрать условия, при которых с олигонуклеотидами будут гибридизоваться только полностью комплементарные фрагменты. Всегда находятся GC-богатые участки, которые будут гибридизоваться и при наличии одного или даже нескольких неспаренных оснований. В связи с этим, метод SBH пока не нашел широкого практического применения. Тем не менее, алгоритмы, разработанные на основе
SBH для сборки коротких прочтений в более длинные фрагменты, стали основой для последующих алгоритмов высокоскоростной сборки и выравнивания, используемых с технологиях секвенирования нового поколения
(new generation sequencing, NGS).

2.4 Секвенирование с помощью масс-спектрометрии MALDI-TOF (Matrix- assistedlaserdesorption/ionization-time-of-flight) (определение нуклеотида по массе и заряду)
 Масс-спектрометрия позволяет идентифицировать компоненты гетерогенной смеси биомолекул по разнице их молекулярных масс. В варианте
Масс-спектрометрия позволяет идентифицировать компоненты гетерогенной смеси биомолекул по разнице их молекулярных масс. В варианте
MALDI-TOF, образец для анализа помещают на поглощающую УФ-излучение подложку и подвергают воздействию короткого лазерного импульса.
Ионизированные молекулы летят в электрическом поле в направлении детектора, причем время, за которое частица достигает детектора обратно пропорционально отношению масса/заряд.
Для определения последовательности нуклеотидов ДНК методом
MALDI-TOF гомогенный фрагмент ДНК (или РНК) высушивают на поверхности в среде 3-гидроксипиколиновой кислоты. ДНК обрабатывают коротким импульсом УФ-лазера, в результате чего ионы ДНК переходят в газовую фазу.
Рис. 24 Принцип секвенирования ДНК на основе метода MALDI-TOF
Заряженные молекулы ДНК в газовой фазе под действием высокого напряжения ускоряются в электрическом поле и попадают на детектор. Затем проводят повторный раунд расщепления и определения масс более мелких
фрагментов. На основании полученных данных может быть вычислена масса анализируемой молекулы и расшифрована последовательность сравнительно короткого гомогенного фрагмента (Рис. 24).
В настоящее время, метод MALDI-TOF не используется в коммерческих вариантах секвенаторов.
2.5 Секвенирование лигированием (принцип комплементарности цепей ДНК)
 Современные методы секвенирования лигированием основаны на использовании коллекции коротких (как правило от 8 до 10 оснований)
Современные методы секвенирования лигированием основаны на использовании коллекции коротких (как правило от 8 до 10 оснований)
флуоресцентно-меченных с помощью четырех красителей вырожденных олигонуклеотидов, так, что каждому флуорофору соответствует определенный нуклеотид (или два) в определенной позиции.
Сначала создают иммобилизованную на твердой фазе клональную библиотеку одноцепочечных фрагментов ДНК (например методом капельной ПЦР). Секвенирование начинают с отжига праймера, комплементарного адаптеру на одном из концов библиотеки ДНК. Затем к библиотеке добавляют флуоресцентно-меченные вырожденные олигонуклеотиды и проводят реакцию лигирования, что приводит к фиксированию олигонуклеотида на фрагменте в случае его полного соответствия. Затем считывают флуоресценцию, определяя тем самым, какой нуклеотид (или пара нуклеотидов) находится в определенной позиции. Флуорофор удаляют и лигируют следующий олигонуклеотид (всего проводят 10 - 15 последовательных лигирований). Затем проводят
"перезагрузку" праймера путем его отсоединения с прилигированными меченными олигонуклеотидами и повторяют цикл с другим праймером со сдвигом на одну букву (Рис. 25).

Рис. 25 Секвенирование методом лигирования
В настоящее время, данный принцип секвенирования реализуется в коммерческих технологиях Prolonator (Dover/Harvard) и SOLiD (Life Technologies Thermo Fisher Scientific).
2.6 Пиросеквенирование (регистрация акта присоединения нуклеотида по образующемуся пирофосфату)
 В 1996 г. специалистами Королевского технологического института в Стокгольме Мустафой Рональди и Полом Ниреном был предложен подход к секвенированию ДНК, в основе которого лежит принцип регистрации пирофосфата, образующегося в результате присоединения очередного
В 1996 г. специалистами Королевского технологического института в Стокгольме Мустафой Рональди и Полом Ниреном был предложен подход к секвенированию ДНК, в основе которого лежит принцип регистрации пирофосфата, образующегося в результате присоединения очередного

нуклеотида ДНК-полимеразой. Для детекции выделяющегося в процессе образования фосфодиэфирной связи пирофосфата используется каскад последовательных химических реакций, заканчивающийся высвечиванием кванта света.
Вначале любым методом создают иммобилизованную на твердой фазе клональную библиотеку одноцепочечных фрагментов ДНК (например, методом мостиковой ПЦР). Предварительно, ко всем фрагментам ДНК присоединяют адаптер, на который будет гибридизоваться праймер, служащий затравкой для синтеза комплементарной цепи ДНК-полимеразой. Дальнейшая реакция состоит из последовательных циклов, в процессе которых, к закрепленной на твердой фазе ДНК по очереди добавляют dNTP всех 4-х типов. В случае, если в данной ДНК-колонии на секвенируемой цепи ДНК имеется комплементарный добавленному нуклеотид, в процессе формирования ДНК-полимеразой фосфодиэфирной связи побочным продуктом реакции будет пирофосфат. Он активирует каскад химических реакций, в результате которых возникает световой сигнал, интенсивность которого прямо пропорциональна числу включенных в цепь нуклеотидов.
Рис. 26 Принцип метода пиросеквенирования
Ферментативные реакции осуществляются АТФ-сульфурилазой,
люциферазой и апиразой. Также в месте с ними в ячейке присутствуют аденозинфосфосульфат (APS) и люциферин. Выделяющийся в ходе образования очередной фосфодиэфирной связи пирофосфат вступает в реакцию
сAPS, катализируемую АТФ-сульфурилазой, с образованием АТФ.
Образовавшийся АТФ является источником энергии для люциферазной
реакции окисления люциферина в оксилюциферин, в процессе которой генерируются кванты света в видимой области спектра, в количестве,
пропорциональном количеству включенных в растущую цепь ДНК нуклеотидов. Световой сигнал регистрируется ПЗС-матрицей (подобной тем,
которые используются в обычных цифровых фотоаппаратах) и анализируется при помощи программного обеспечения, преобразующего так называемую пирограмму в последовательность нуклеотидов (Рис. 26).
Не вовлеченные в синтез новой цепи нуклеотиды, а также АТФ деградируются при помощи апиразы. После этого можно начинать следующий цикл, т.е. добавлять другой тип нуклеотида. На принципе пиросеквенирования основана коммерческая технология 454 Life Sciences Roche.
2.7 Метод обратимых терминирующих нуклеотидов (регистрация каждого присоединенного нуклеотида по отщепляемой метке)
 Данная концепция была предложена Баласубрамяном и Кленерманом на химическом факультете Кембриджского университета. Также как и при пиросеквенировании, принцип метода состоит в регистрации факта присоединения очередного нуклеотида, но не по побочным продуктам реакции,
Данная концепция была предложена Баласубрамяном и Кленерманом на химическом факультете Кембриджского университета. Также как и при пиросеквенировании, принцип метода состоит в регистрации факта присоединения очередного нуклеотида, но не по побочным продуктам реакции,
а непосредственно по сигналу от присоединенного основания. При этом должны выполняться два требования:
1) за один цикл реакции может быть добавлен только один нуклеотид.
Это легко обеспечить с использованием 3ʹ-блокированных dNTP с
возможностью снятия блока.
2) метка должна быть отщепляемой.
Сначала любым методом создают иммобилизованную на твердой фазе клональную библиотеку одноцепочечных фрагментов ДНК (например, методом мостиковой или эмульсионной ПЦР). Секвенирование начинают с отжига праймера, комплементарного адаптеру на одном из концов библиотеки ДНК.
Затем к библиотеке добавляют четыре типа флуоресцентно меченых обратимых терминирующих dNTP (так называемых RT-оснований). ДНК-полимераза
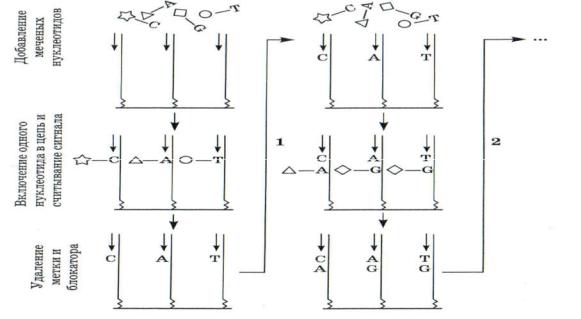
присоединяет подходящий нуклеотид к затравке и на этом синтез временно
останавливается.
Рис. 27 Принцип секвенирования синтезом клональной библиотеки одноцепочечных
фрагментов ДНК на твердой фазе
Невключившиеся нуклеотиды смывают, и оптическая система считывает флуоресценцию каждой ДНК-колонии библиотеки. Каждая колония высвечивает при этом кванты флуоресценции, соответствующие включившемуся на данном этапе нуклеотиду. После этого флуорофор, наряду с
3ʹ-концевым блокатором, химически удаляют из синтезированной цепи, что позволяет повторить весь цикл сначала (Рис. 27).
В отличие от пиросеквенирования в данном подходе сигнал флуоресценции можно регистрировать в течение длительного времени после присоединения очередного нуклеотида, что позволяет производить анализ очень большого количества ДНК-колоний.
На данном принципе секвенирования основаны коммерческие технологии компаний Illumina и Pacific Bioscience.
2.8 Полупроводниковое секвенирование (регистрация акта присоединения нуклеотида по образующимся ионам водорода)
 Полупроводниковое секвенирование - это метод определения последовательности ДНК, основанный на детекции ионов водорода,
Полупроводниковое секвенирование - это метод определения последовательности ДНК, основанный на детекции ионов водорода,
